1月27日,清华大学化工系生物育种技术与装备团队在《核酸研究》(Nucleic Acids Research)发表文章,采用高通量手段系统研究了sgRNA与靶标DNA错配效应对dCas9结合活性的影响。
近年来,作为可编程的DNA靶向工具,CRISPR/(d)Cas系统在基因编辑、高通量遗传筛选、染色体成像、合成基因路线构建、新型抗菌药物和体外核酸检测等领域得到了广泛的应用。对于每个应用而言,向导RNA(sgRNA)与靶标DNA之间的序列同源性对CRISPR/(d)Cas系统活性的影响都是至关重要的。为加深对这个问题的理解,作者在大肠杆菌中构建了两个覆盖所有单核苷酸和双核苷酸突变的sgRNA文库,并利用高通量的方法在活细胞内解析了文库中每个sgRNA介导dCas9与靶标DNA结合的亲和力。
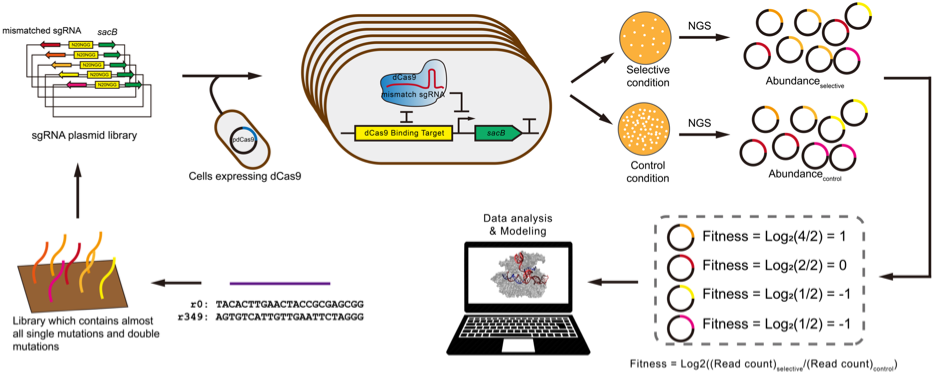
图1 高通量解析不同错配sgRNA对dCas9与靶标结合活性的影响
该研究基于高通量实验手段发现种子区域(PAM近邻7-12 bp)的错配对结合活性的影响较大,而PAM远端区域可以容忍多个错配;种子区的错配存在协同效应,即两个错配的组合对结合活性的影响大于两个对应单独错配影响的加和。特别的,相对于其它错配类型,dDrG(D = A, T, G)对dCas9结合的影响相对温和,与核酸热力学数据呈现出一致性,并得到了进一步的实验验证。
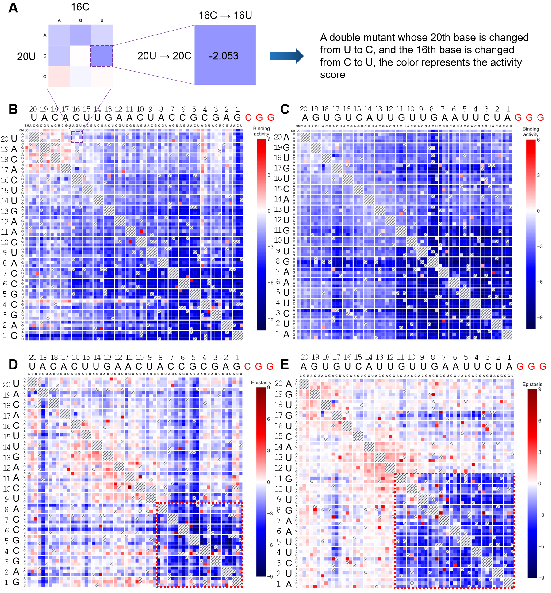
图2 错配sgRNA介导的CRISPR/dCas9结合活性的系统解析
CRISPR/(d)Cas9识别并结合靶标主要分为两个步骤,首先是PAM (NGG)的识别及其近邻碱基的熔化,接着是链侵入过程,即sgRNA与熔化的靶标DNA发生退火,并延伸至PAM远端。基于CRISPR/(d)Cas9的结合机理和实验观测,论文将链侵入过程的状态转移视作马尔可夫链,并使用文献报道的核酸热力学数据计算每个状态的概率,最终发现dCas9的解离概率与结合亲和力存在较强的负相关关系,并能很好的与文献报道的体外实验数据吻合。该模型也能外推至更多错配的情形,一定程度上证明了其普适性,对于深入理解和应用CRISPR系统的脱靶结合效应具有重要意义。
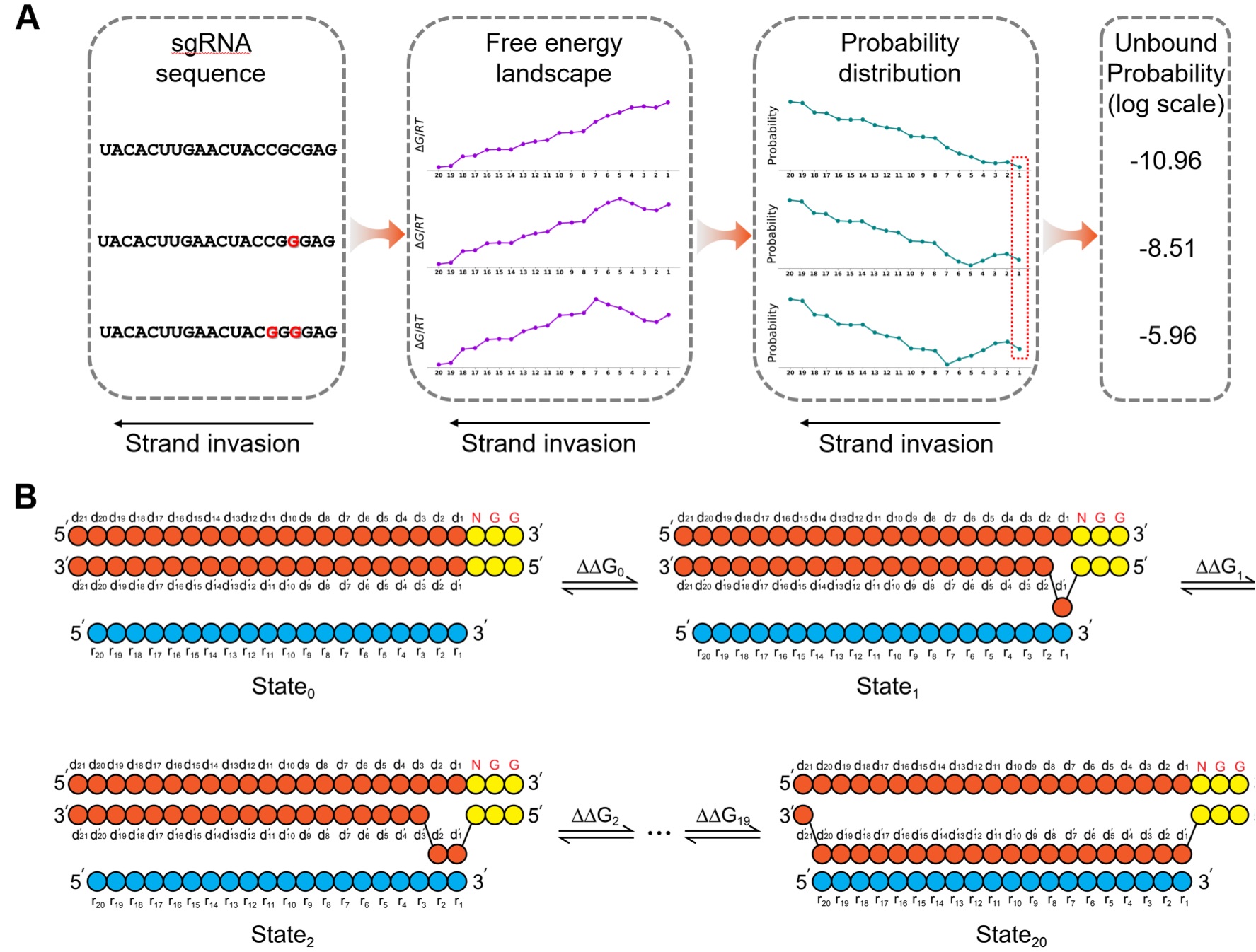
图3 CRISPR/dCas9的结合亲和力受核酸热力学控制
化工系张翀副教授和王天民博士(现为清华大学医学院博士后)为本文通讯作者,生物育种技术与装备团队首席邢新会教授为本文共同作者。冯汇宝博士研究生、郭佳荟博士、王天民博士为本文的共同第一作者。该成果得到了国家自然科学基金委重点仪器研发项目、面上项目和博士后基金的资助。
论文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkaa1295/6121456


 微信公众号
微信公众号 
